COVID-19疫情简要可视化分析
2020年全球遭遇了新冠肺炎疫情,各大门户网站和主流App其实都有多维度的疫情数据分析。但是我还是打算尝试做一些简单的数据分析展示,同时会将数据以地图的形式可视化展示。
整个数据文件一共有两个分别为data_ncov.xlsx和chinadata.json。前一个文件是我们的疫情数据集文件,后一个则是后续创建地图时用到的全国各地省市的地理数据信息。
# COVID-19 数据分析
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import warnings
# 设置不弹出警告
warnings.filterwarnings('ignore')
# 中文乱码设置
plt.rcParams['font.sans-serif']=['SimHei']
plt.rcParams['axes.unicode_minus']=False
# retian屏幕显示设置
%config InlineBackend.figure_format = 'retina'
# 读取数据
url = '/Users/Anders/Documents/Jupyter/DataSet/COVID-19/data_ncov.xlsx'
# 数据集文件格式为xlsx,故使用read_excel方法读取文件
df = pd.read_excel(url)
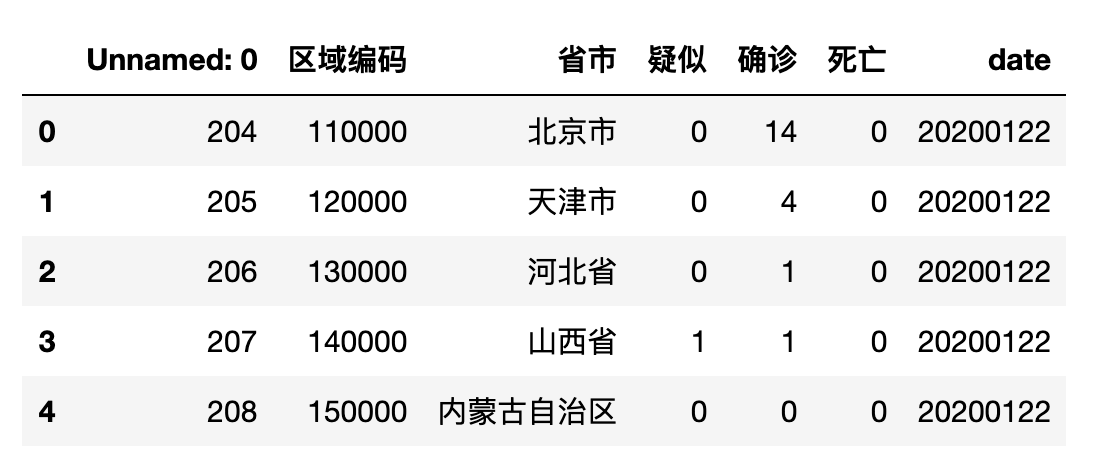
df.head()
先读取数据集文件并输出前5条数据内容格式如下:
在进行基本的数据清洗前,可以通过info方法先了解数据的基本信息,如字段类型、数据一致性、是否存在空值等。
df.info()
输出如下:
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 782 entries, 0 to 781
Data columns (total 7 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Unnamed: 0 782 non-null int64
1 区域编码 782 non-null int64
2 省市 782 non-null object
3 疑似 782 non-null int64
4 确诊 782 non-null int64
5 死亡 782 non-null int64
6 date 782 non-null int64
dtypes: int64(6), object(1)
memory usage: 42.9+ KB
从如上输出可以发现date字段为int64类型,并不是日期类型,考虑到后续需要与日期字段打交道,为了更方便比较,这里选择先将该字段转化为字符串类型更合适。
# 当前数据日期信息的类型为整型,所以先将字段转化为字符串
df['date'] = df['date'].astype('str')
df['date'] = pd.to_datetime(df['date'])
一、各省市疫情排行
为了查看各省市的疫情数据排行,编写一个函数可以方便查看(除了湖北省以外)10大省市排行最高的确诊类型数据,该函数参数支持自定义,如指定时间、类型、几大排行。
def fig_top_type(time, ty, top_num):
# 根据传入的时间参数'time'筛选指定时间日期。
df_data = df[df['date']==time]
# 根据传入的指定类型'ty'排序,按照从大到小顺序排序,并修改原数据。
df_data.sort_values(by=ty, ascending=False, inplace=True)
# 因为考虑到当前数据湖北省远远领先,只取湖北省外的数据所以切片从1开始
df_data.iloc[1:top_num+1].plot(x = '省市', y=ty,
kind='bar', figsize=(15,5),
grid=True, rot=45,
title='湖北省外{0}日,{1}病例最多的{2}省市排行'.format(time, ty, top_num))
# 输出一个除了湖北省外2020年2月1日关于确诊和疑似的柱状图
fig_top_type('20200201',['确诊','疑似'], 10);
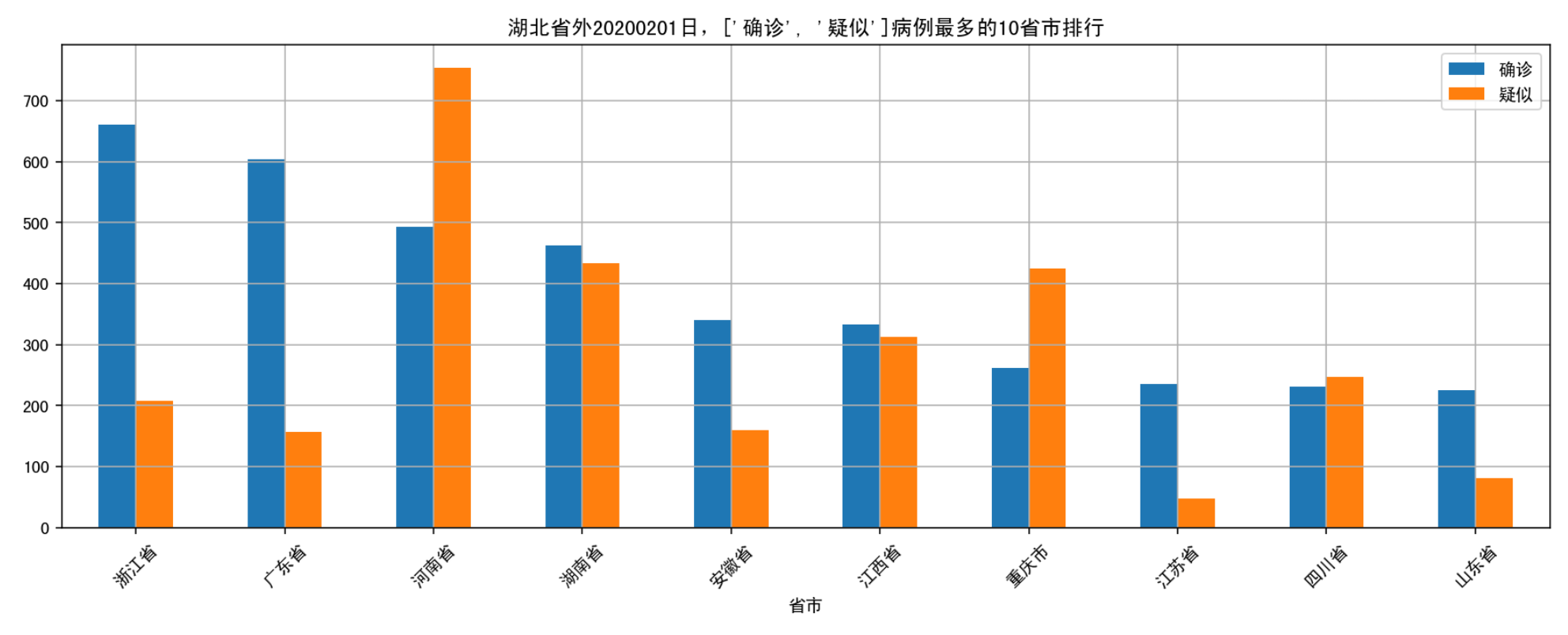
这里很清楚的可以看到2020年2月1日排名前三的分别是浙江省、广东省、河南省。
二、(全国) 每日病例与增长率数据展示
之前我们以省市为单位展示了数据排行的情况,但是如果想知道全国每天的新增数据情况呢?
由于目前提供的数据都是每个省市每一天的累积更新,想获得每天新增的各类病例,就需要计算得到。
既然以每天的日期为单位,就可以使用groupby针对日期进行分组,把所有城市每天的疫情数据汇总起来,按照 "疑似"、"确诊"、"死亡"三类进行分类分组并汇总求和。
# 以日期分组,获取全国每天的 疑似、确诊、死亡 数据。
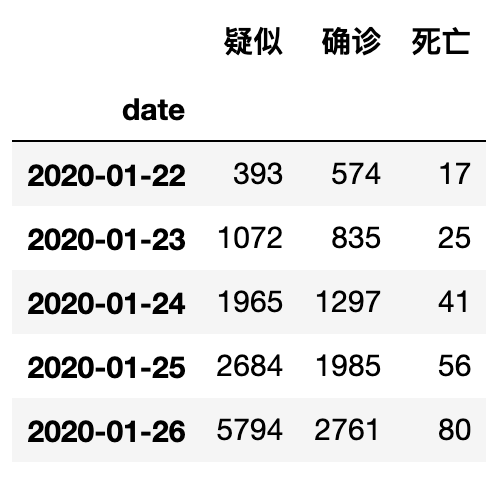
data_china = df.groupby('date')[['疑似','确诊','死亡']].sum()
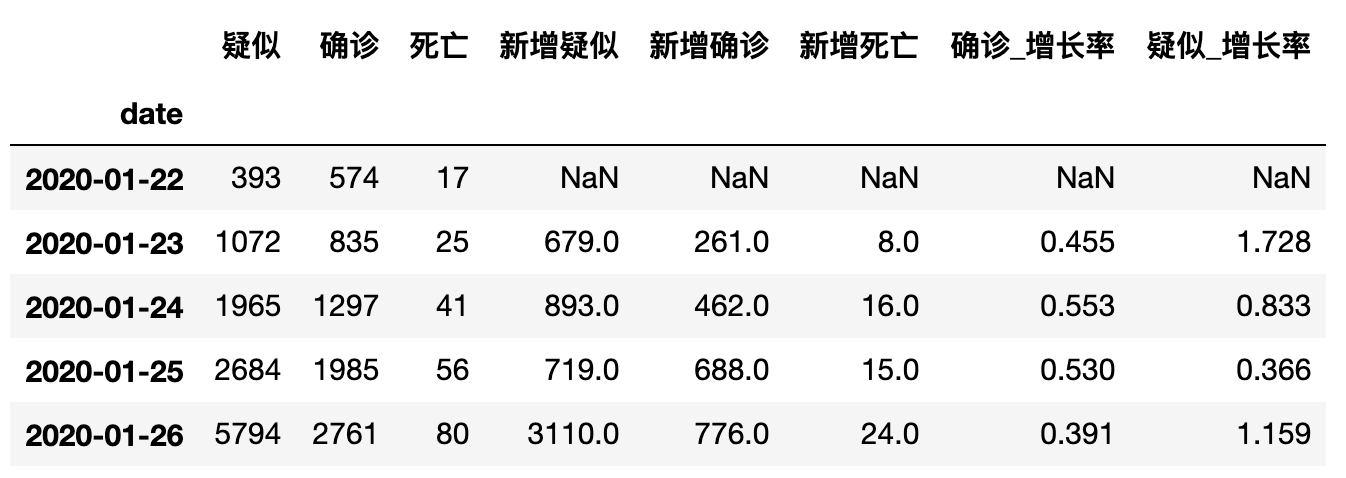
data_china.head()
输出如下:
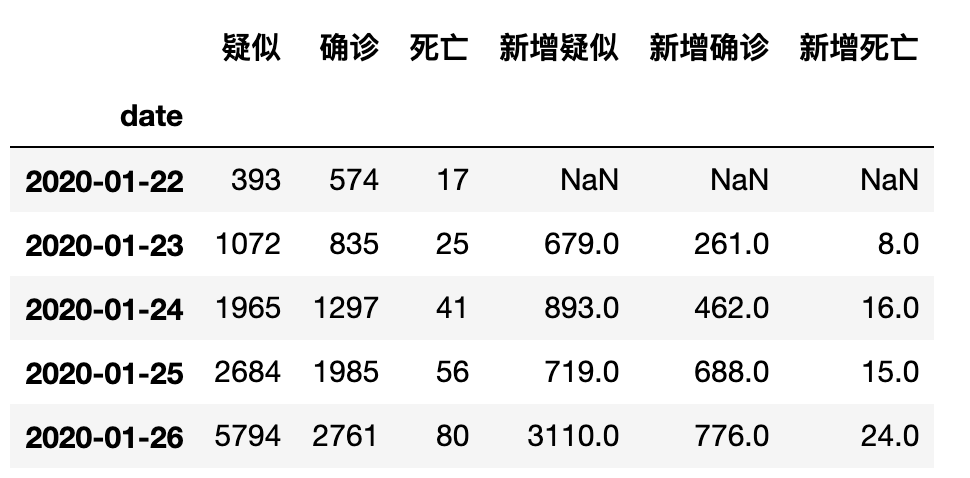
因为通过观察得知每天的原始数据是累积的数据,所以要得到每天各类的新增数据就必须拿后一天的数据减去前一天数据,那么使用shift方法可以使数据进行前移或者后移,其中shift(1)代表向下平移1位,shift(-1)代表向上平移一位。同时我们添加【新增**】列字段保存每日新增的数据。
# 为了计算后一日对前一日的数据差计算,使用shift方法可以用于同行单纯的前移或后移操作
data_china['新增疑似'] = data_china['疑似'] - data_china['疑似'].shift(1)
data_china['新增确诊'] = data_china['确诊'] - data_china['确诊'].shift(1)
data_china['新增死亡'] = data_china['死亡'] - data_china['死亡'].shift(1)
data_china.head()
有了各项新增数据后,也可以添加更直观的增长率(百分比)。比如确诊的增长率就等于用当天的新增确诊数据 除以 前一日的确诊数据就代表当天的确诊增长率。由于直接使用新增确诊除以确诊在这里除以的是相同行内当天的确诊数据,所以我们必须使用shift使数据向下移动一位。
# 计算新增确诊的增长率就等于用新增的数据除以新增前的数据。
data_china['确诊_增长率'] = data_china['新增确诊'] / data_china['确诊'].shift(1)
data_china['确诊_增长率'] = data_china['确诊_增长率'].round(3)
data_china['疑似_增长率'] = data_china['新增疑似'] / data_china['疑似'].shift(1)
data_china['疑似_增长率'] = data_china['疑似_增长率'].round(3)
data_china.head()
基本的数据清理完毕后,我们打算开始进行可视化,这里选择使用百度的Echarts做图形可视化。
# https://pyecharts.org/#/zh-cn/intro
# Echarts 是一个由百度开源的数据可视化,而pyecharts是封装了Echarts的python库,
# 为了使用python能更方便的使用Echarts图形。注:新版本V1以后版本要使用新的调用方法。
from pyecharts.charts import Bar, Grid, Line
from pyecharts import options as opts
from pyecharts.globals import ThemeType
# x轴为日期为单位分类。考虑到第一行数据本身不存在新增对比,数据存在Nan值,所以我们舍弃第1行,当然如果需要也可以做合理的填充处理。
x = data_china.iloc[1:].index.astype('str')
y1 = data_china[['确诊','疑似']].iloc[1:]
bar = (
Bar(init_opts=opts.InitOpts(theme=ThemeType.INFOGRAPHIC, width="700px", height="350px"))
.add_xaxis(list(x))
.add_yaxis('确诊',list(y1['确诊']))
.add_yaxis('疑似',list(y1['疑似']), gap='10%')
.extend_axis(yaxis=opts.AxisOpts())
.set_series_opts(label_opts=opts.LabelOpts(is_show=False))
.set_global_opts(title_opts=opts.TitleOpts(title="全国累计确诊病例 柱状图", title_textstyle_opts=opts.TextStyleOpts(font_size=15)),
datazoom_opts=opts.DataZoomOpts(),
tooltip_opts = opts.TooltipOpts(axis_pointer_type='cross'))
)
y2 = data_china['确诊_增长率'].iloc[1:]
y3 = data_china['疑似_增长率'].iloc[1:]
line = (
Line()
.add_xaxis(x)
.add_yaxis('确诊增长率', y2, yaxis_index=1, is_smooth=True)
.add_yaxis('疑似增长率', y3, yaxis_index=1, is_smooth=True)
.set_series_opts(label_opts=opts.LabelOpts(is_show=False))
)
bar.overlap(line)
bar.render_notebook()
使用Echarts可视化后输出的图形可以很方便的进行数据的交互。
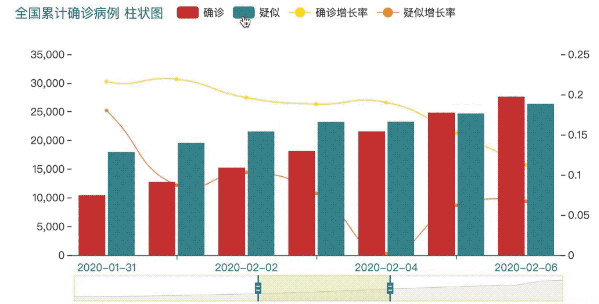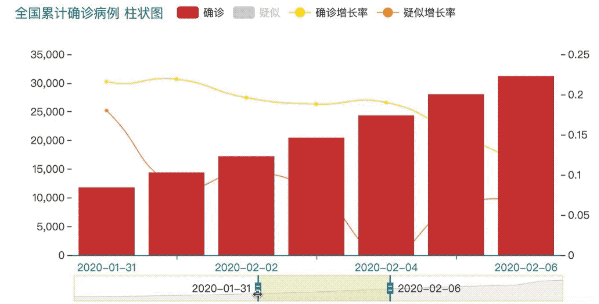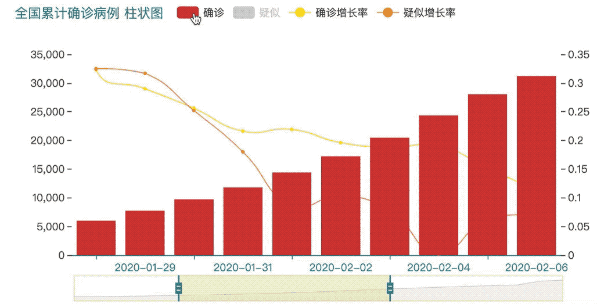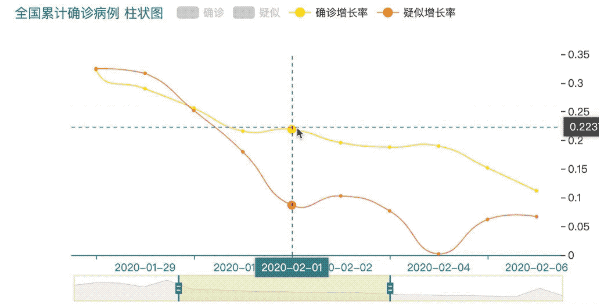
三、中国疫情地图
用地图展现疫情数据可以更直观的从地域上来了解疫情的严重程度的分布情况。当然地图的可视化方法有很多,这里我们分别使用GeoPandas库和Echarts包内自带的Map库制作。
- 使用GeoPandas库实现可视化
# GeoPandas是一个开源项目,它的目的是使得在Python下更方便的处理地理空间数据。
# GeoPandas扩展了pandas的数据类型,允许其在几何类型上进行空间操作。
import geopandas as gpd
geo_url = '/Users/Anders/Documents/Jupyter/DataSet/COVID-19/chinadata.json'
# 通过geopandas.GeoDataFrame.from_file()方法来读取数据,对于空间数据的格式常见的为*.json与.shapefile(GIS类软件常用)
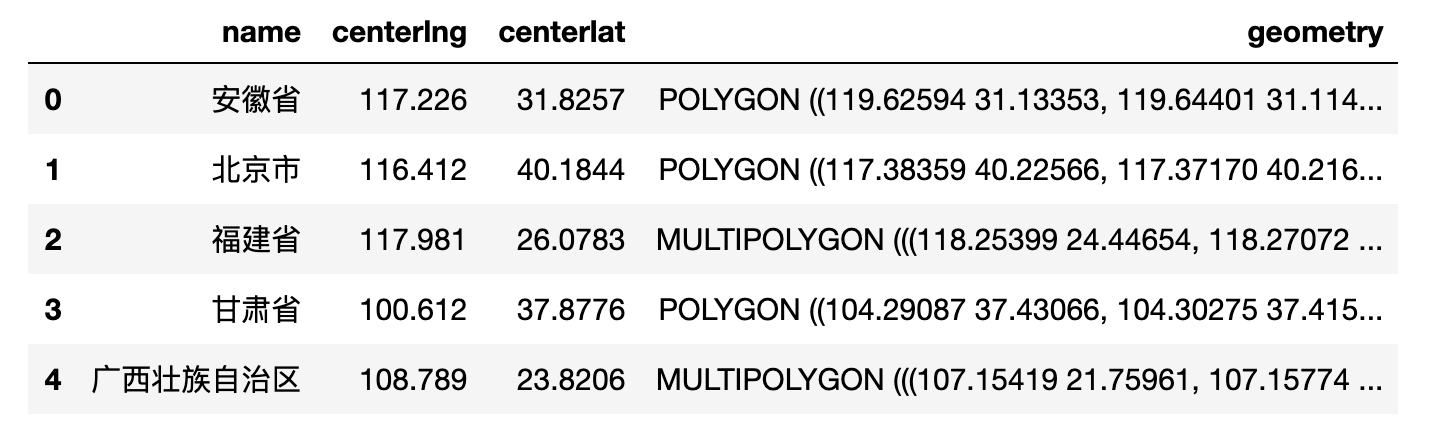
china_spatial = gpd.GeoDataFrame.from_file(geo_url)
china_spatial.info()
读取chinadata.json文件后使用info方法查看信息输出如下。
<class 'geopandas.geodataframe.GeoDataFrame'>
RangeIndex: 34 entries, 0 to 33
Data columns (total 4 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 name 34 non-null object
1 centerlng 34 non-null float64
2 centerlat 34 non-null float64
3 geometry 34 non-null geometry
dtypes: float64(2), geometry(1), object(1)
memory usage: 1.2+ KB
可以看到geometry字段数据类型为geopandas.geodataframe.GeoDataFrame,并不是pandas读取后的pandas.DataFrame,但由于geopandas结合了pandas和shapely的功能,所以对于表格的处理方法基本和pandas一致。
可以这样理解,GeoDataFrame是向DataFrame增加了地理数据支持的功能。我们也能发现新的数据字段类型:geometry,该类型字段代表的是空间信息,比如这里的数据其实是我国省级行政区划的面数据,面数据的空间属性为:面中每个节点的经纬度坐标构成。
china_spatial.head()
通过head方法查看提取的chinadata.json前几行内容如下。
为了让地图里每个省市有自己的对应数据,我们就需要把疫情数据和对应的省市匹配起来。假设我们需要用地图的方式显示2020年2月1日的疫情数据,那么我们
data_0201 = df[df['date'] == '2020-2-1']
data_0201.head()
接下来开始绘制地图,逻辑分为:先把地理数据与疫情数据合并,接着创建画布,然后使用合并后带有地理坐标的疫情数据集去结合plot方法绘制,最后是借助plt.text文本方法给地图上的每个地理位置标注省市的名称。
# 把地图数据与疫情数据匹配起来
data_china_0201 = pd.merge(china_spatial, data_0201, left_on = 'name', right_on = '省市', how = 'left')
data_china_0201.drop(['name'], axis=1, inplace=True)
data_china_0201.head()
# 创建画布
plt.figure(figsize=(12,9))
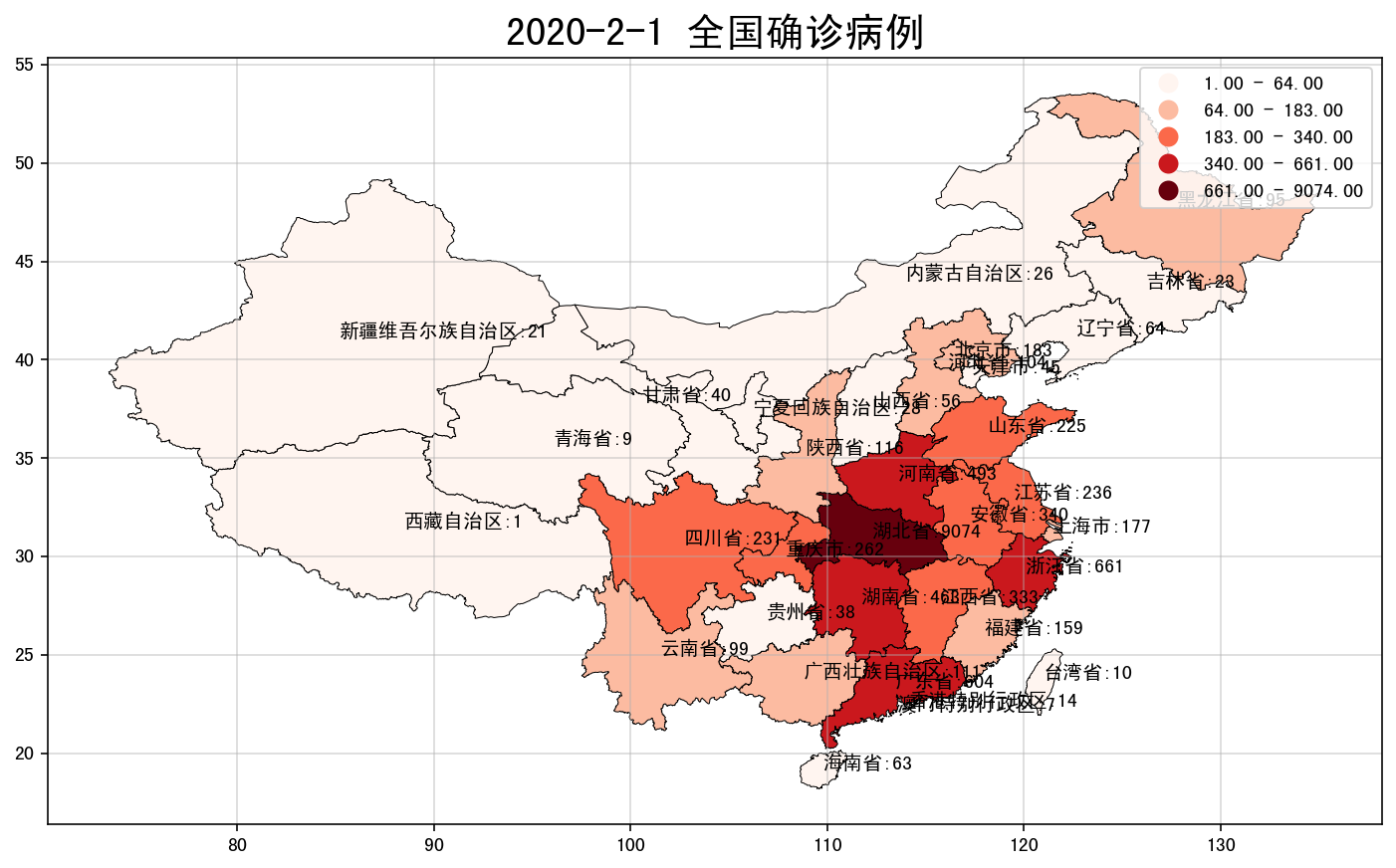
plt.title('2020-2-1 全国确诊病例', fontsize = 20)
# 绘制疫情地图,这里我们只显示确诊信息,所以设置参数column='确诊'
data_china_0201.plot(ax=plt.subplot(1,1,1), alpha=1, edgecolor='k', linewidth = 0.5,
legend=True, scheme = 'FisherJenks', column='确诊', cmap = 'Reds')
# 设置网格线
plt.grid(True,alpha=0.5)
# 添加省市信息
lst = data_china_0201[['省市','centerlng','centerlat','确诊']].to_dict(orient = 'record')
for i in lst:
plt.text(i['centerlng'], i['centerlat'], i['省市'] +':' + str(i['确诊']))
现在就可以很方便的从地图上看到哪个省市到2月1日为止的疫情最为严重,颜色深度越深代表疫情越严重。
除了如上以不同颜色来展示外,还可以在地图中绘制以气泡图的方式来展现。绘图逻辑和上一关大体相同,不过地理空间图将会作为底图,也就不需要指定column参数。这里需要用到matplotlib的scatter方法将xy轴参数去匹配地理坐标的经纬度数据,而其中s参数就是疫情的确诊数据。
# 把地图数据与疫情数据匹配起来
data_china_0201 = pd.merge(china_spatial, data_0201, left_on = 'name', right_on = '省市', how = 'left')
data_china_0201.drop(['name'], axis=1, inplace=True)
plt.figure(figsize=(14,18))
plt.title('2020-2-1 全国确诊病例', fontsize = 23)
# 绘制底图地图时不需要显示一列数据,所以不需要指定column参数,只需要显示一个底图颜色。
data_china_0201.plot(ax=plt.subplot(1,1,1),
edgecolor='k', linewidth = 0.5, color = 'gray', alpha = 0.1)
# 添加气泡图
plt.scatter(data_china_0201['centerlng'], data_china_0201['centerlat'],
s = data_china_0201['确诊'], edgecolors='k', alpha = 0.8)
# 设置网格线
plt.grid(True,alpha=0.5)
# 添加省市信息
lst = data_china_0201[['省市','centerlng','centerlat','确诊']].to_dict(orient = 'record')
for i in lst:
plt.text(i['centerlng'], i['centerlat'], i['省市'] +':' + str(i['确诊']))
如下图,就可以很直观的以气泡的方式发现哪个省市的疫情气泡大就说明疫情更严重。
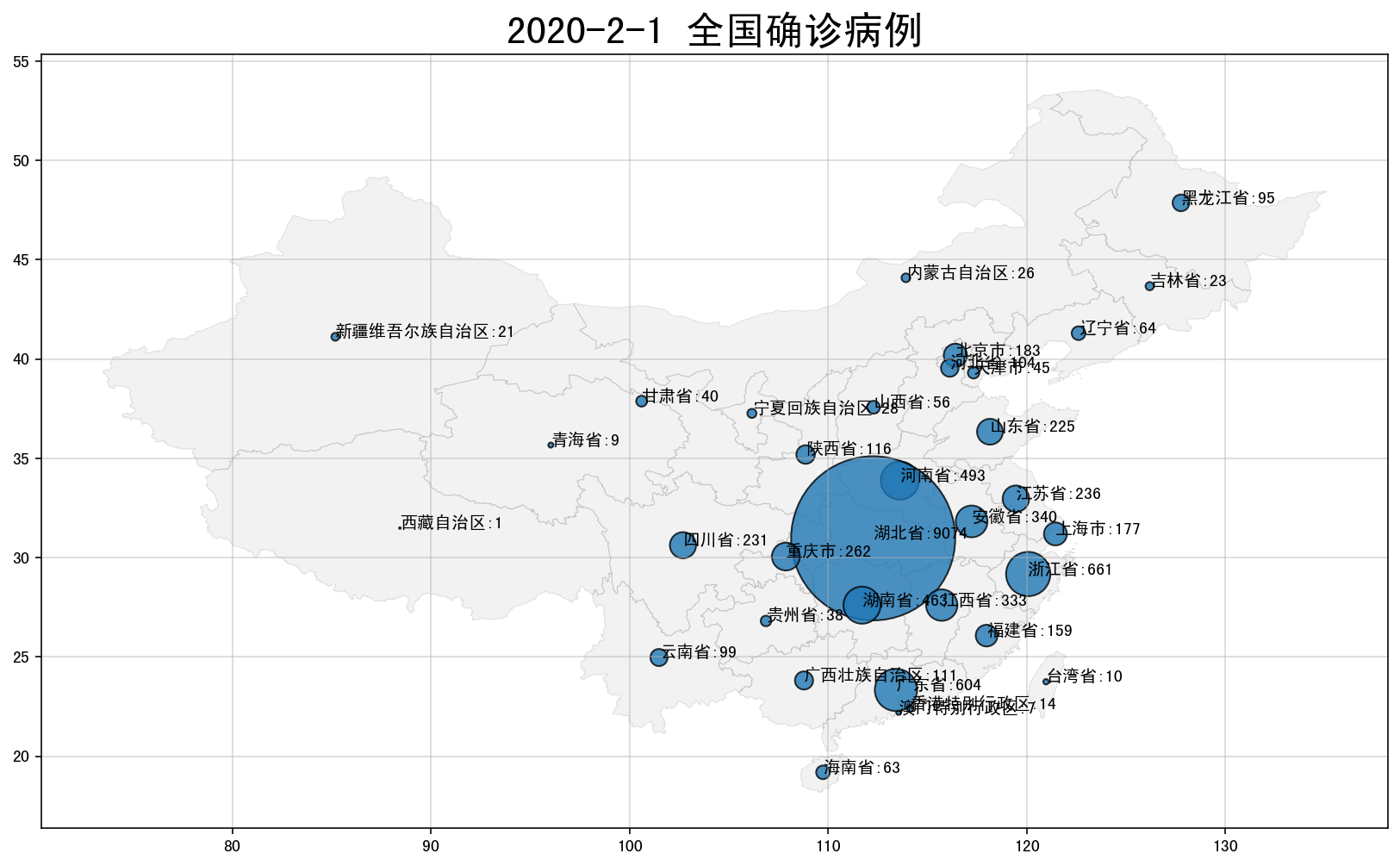
甚至还可以包装成一个函数,批量创建气泡地图,考虑到批量创建,地图显示局促,所以我们取消了地理名称的文本显示。
def create_map(time, tp, x, y, n):
# 按照日期筛选数据
datai = df[df['date'] == time]
# 匹配数据
data_chinai = pd.merge(china_spatial, datai, left_on = 'name', right_on = '省市', how = 'left')
del data_chinai['name'] # 删除多余字段
# 绘制底图
data_chinai.plot(ax=plt.subplot(x,y,n),
edgecolor='k', linewidth = 0.5,
color = 'gray', alpha = 0.1)
# 添加气泡图
plt.scatter(data_chinai['centerlng'],data_chinai['centerlat'],
s = data_chinai[tp], edgecolors='k', alpha = 0.8)
# 设置标题及网格线
plt.title('%s 全国%s病例' % (time, tp), fontsize = 20)
plt.grid(True,alpha=0.5)
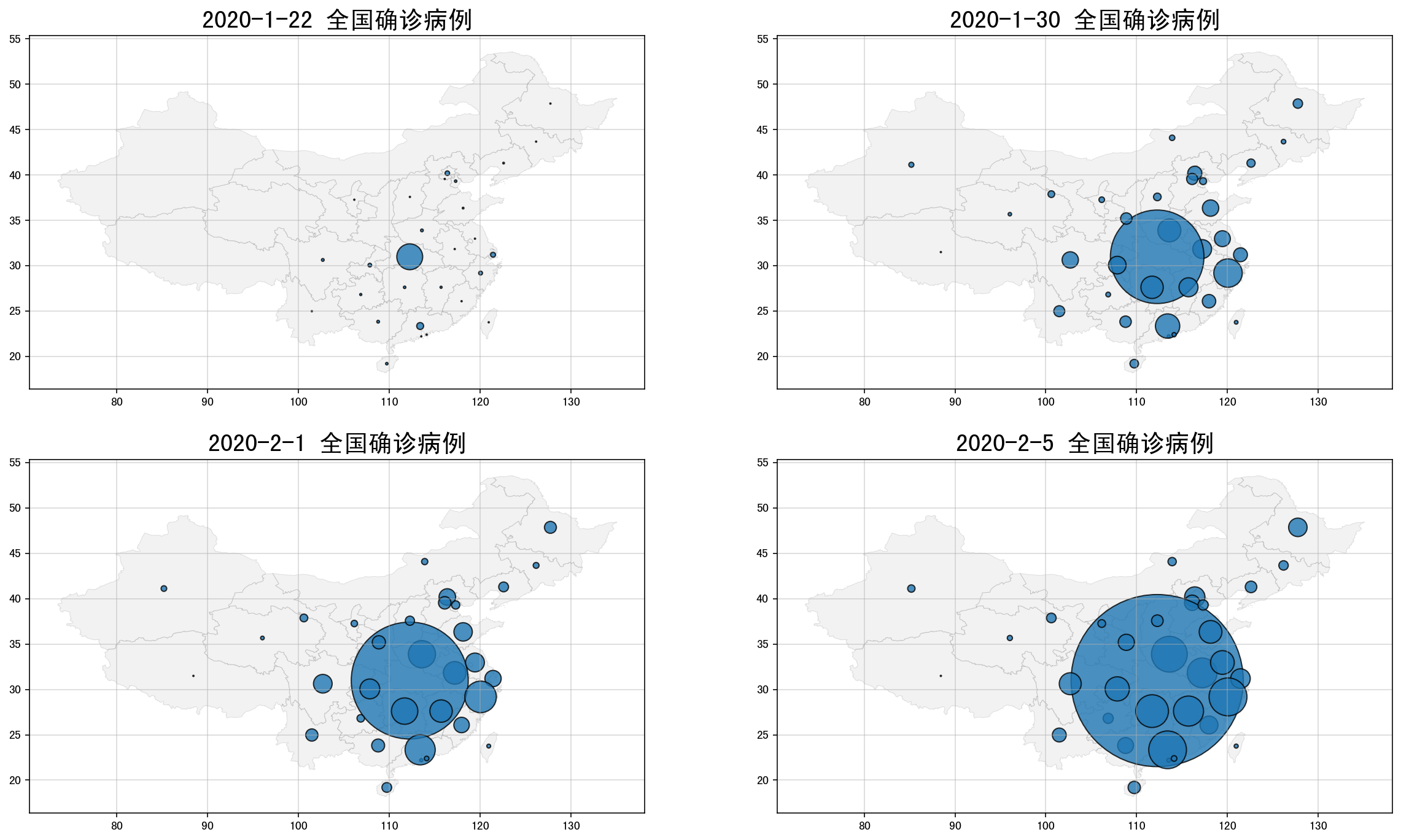
# 构建for循环批量出图
# 设置日期列表
datelst = ['2020-1-22','2020-1-30','2020-2-1','2020-2-5']
# 创建绘图对象
plt.figure(figsize=(20,18))
# 批量出图
m = 1
for i in datelst:
create_map(i,'确诊', 3,2,m)
m += 1
下图批量显示了'2020-1-22','2020-1-30','2020-2-1','2020-2-5'4个时间的疫情气泡大小变化。
- 使用pyecharts库实现可视化
pyecharts库中负责地理坐标系的模块是Geo,负责地图的模块是Map,负责百度地图的模块是BMap。
其中Geo实现了一个地理坐标系,地图上的点可以与经纬度进行转换(即可以利用经纬度向地图中插入点,也可以获取地图上某一点的经纬度),实现地图上的打点功能主要依靠Geo类来进行,而Map功能类似于Geo,但只有地图,没有坐标系,即地图上的点无法与经纬度进行转换。而负责图表配置的模块是options。在 pyecharts 中,图表的一切皆通过 options来修饰调整。
from pyecharts.faker import Faker
from pyecharts import options as opts
from pyecharts.charts import Map
# 整理原先数据集里的地理名称,为了使与pyecharts内的map包内的地理名称所匹配
dict_special_str = {'广西壮族自治区':'广西', '内蒙古自治区':'内蒙古',
'宁夏回族自治区':'宁夏', '西藏自治区':'西藏', '新疆维吾尔族自治区':'新疆'}
special_str_func1 = lambda x: x.replace('市','').replace('省','').replace('特别行政区','')
provinces = data_china_0201['省市'].map(special_str_func1)
def special_str_func2(s):
if dict_special_str.setdefault(s):
return dict_special_str[s]
else:
return s
provinces = list(map(special_str_func2, provinces))
value = list(data_china_0201['确诊'])
def map_base():
c = (
Map()
.add("确诊", [list(z) for z in zip(provinces, value)], "china")
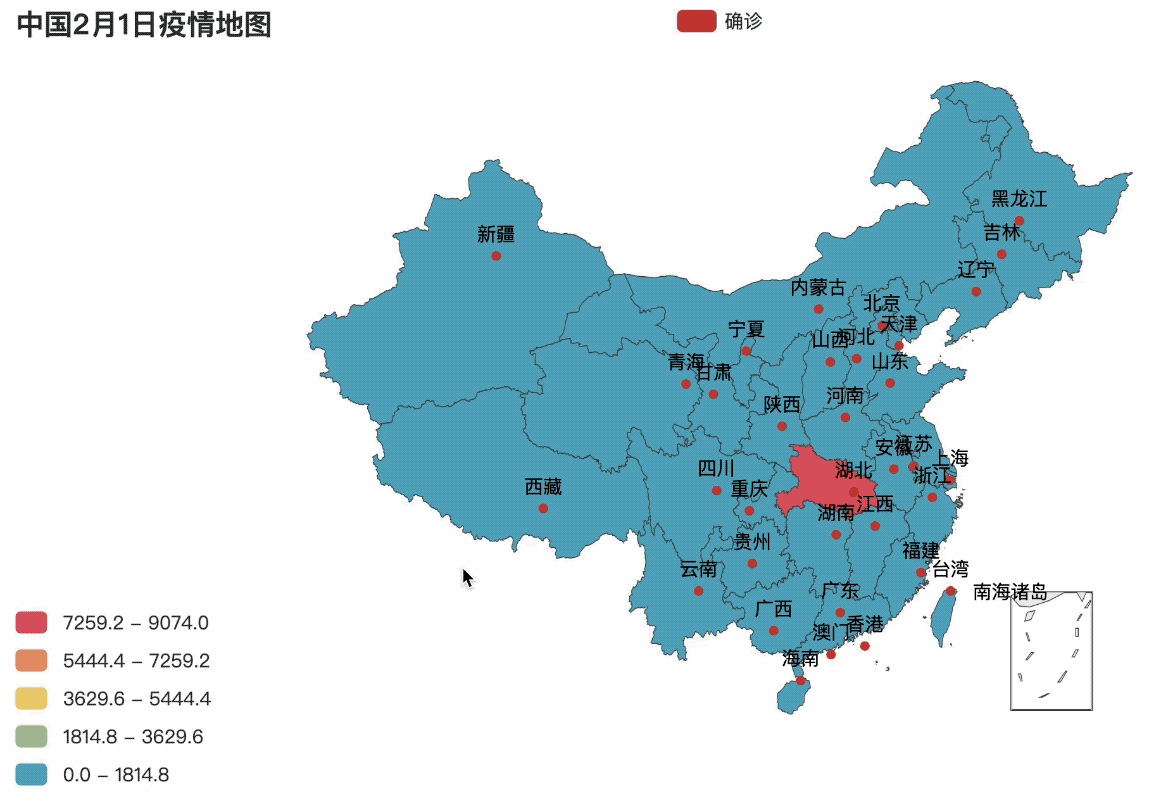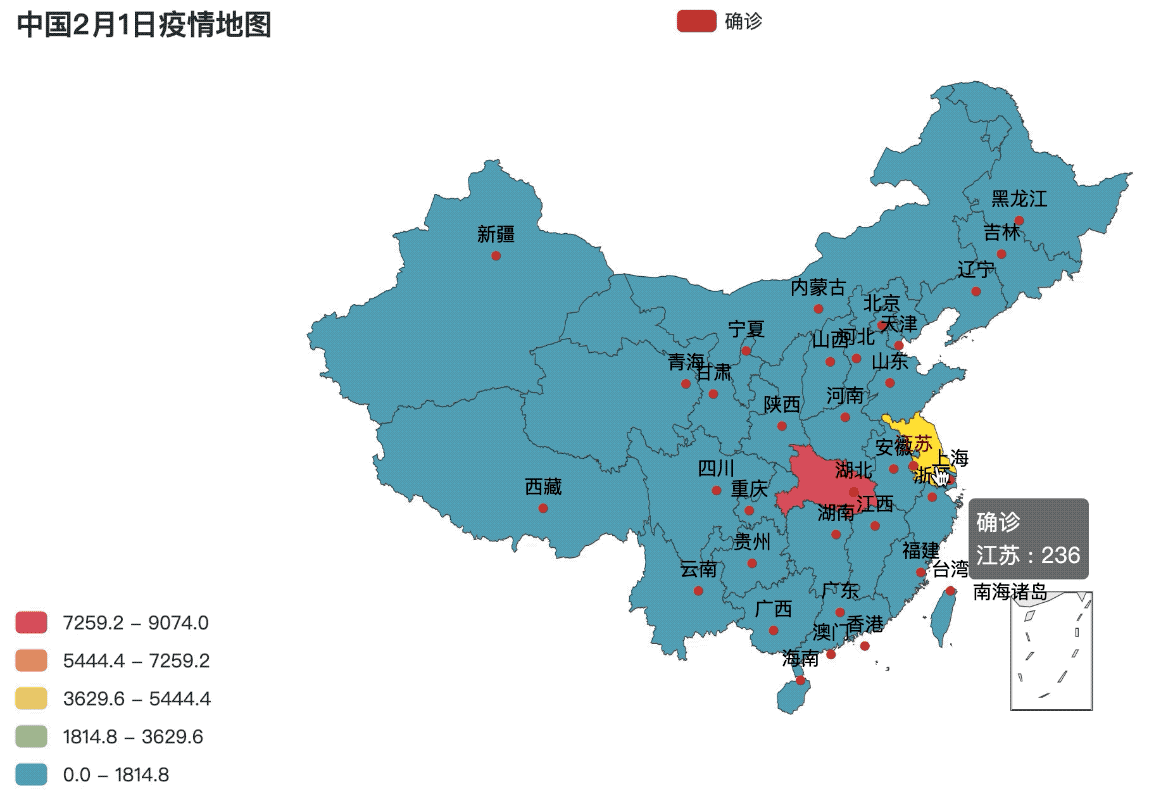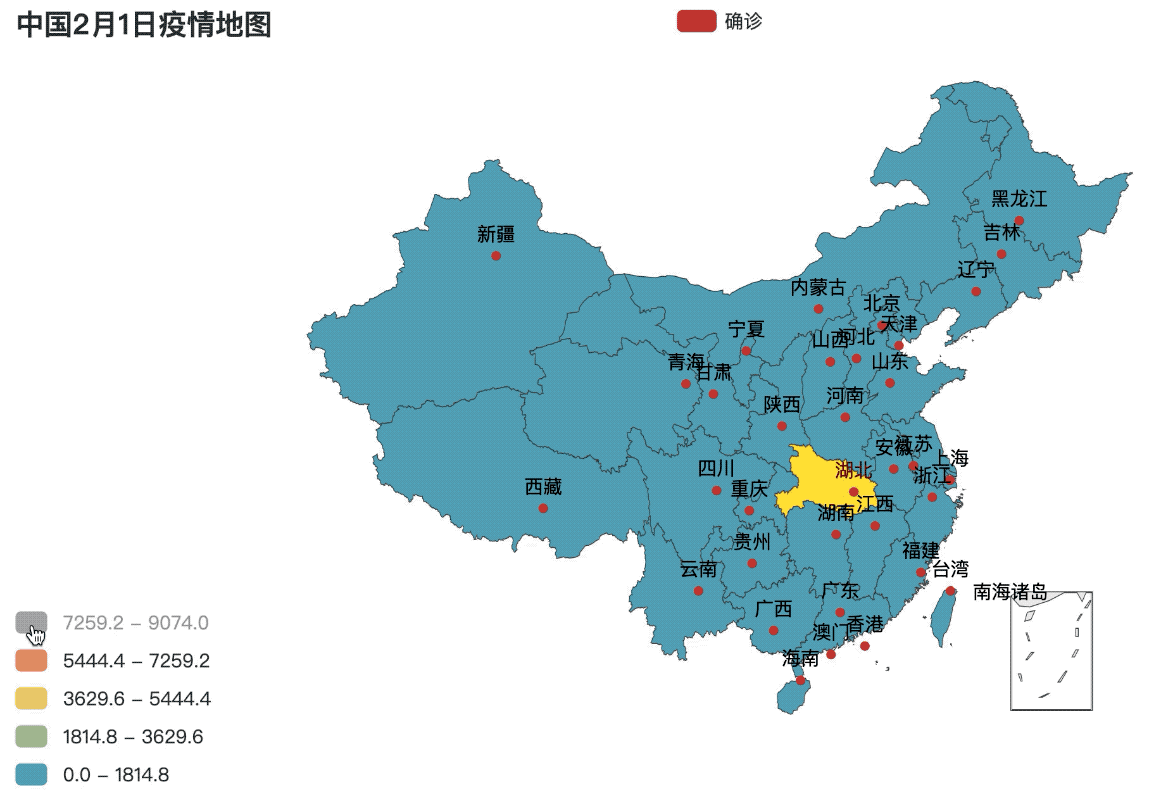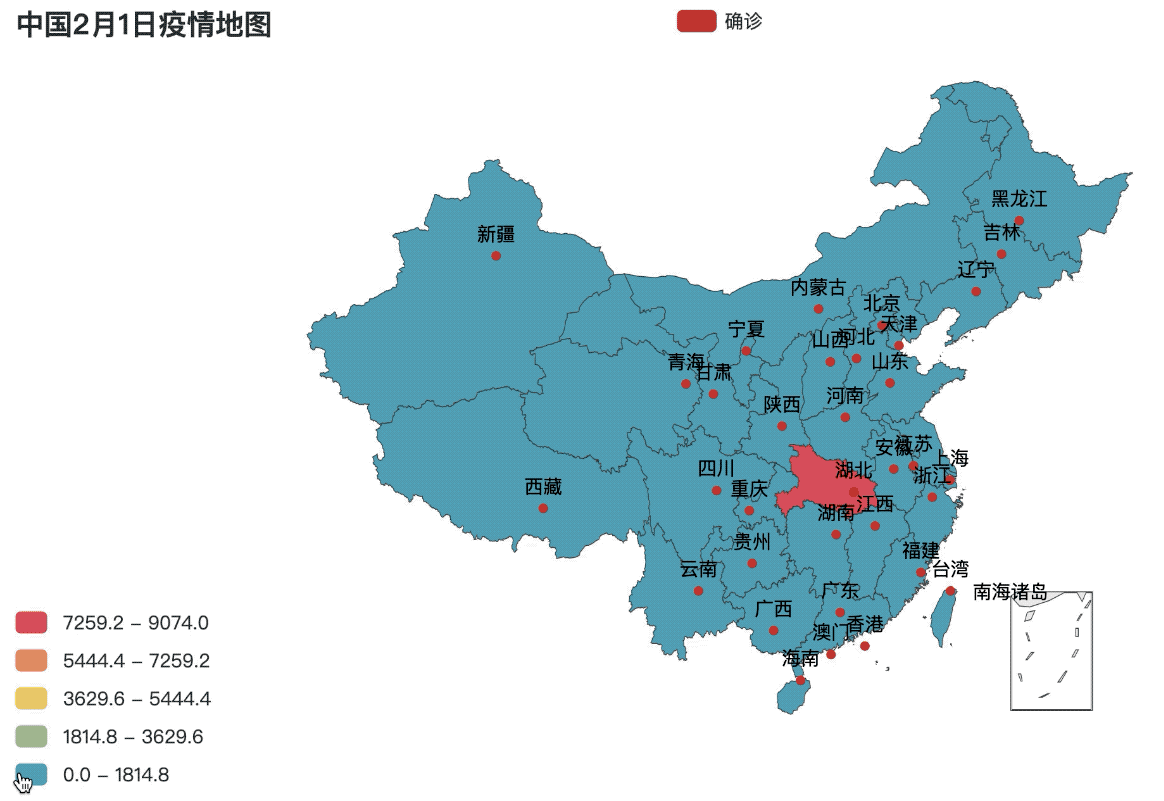
.set_global_opts(title_opts=opts.TitleOpts(title="中国2月1日疫情地图"),
visualmap_opts=opts.VisualMapOpts(is_piecewise=True, max_=max(value), min_=0))
)
return c
city_map = map_base()
city_map.render_notebook()